"Recueil de données"...
1. Préparer les données (DB) : "Data Munging" (ou Data Wrangling)
Récupérer, mélanger, enrichir, filtrer, nettoyer, vérifier, formater, transformer des données...
2. Mettre en oeuvre une méthode un modèle (ML/Stats)
Arbre de décision, régression, clustering, Modèle graphique, SVM...
3. Interpréter les résultats : visualisation
Graphiques, Data visualisation, Cartes...
Base : le fichier texte
Format simple et pérenne pour stocker des données et les échanger.
Exemples :
- CSV : Comma Separated Value
- XML : Extensible Markup Language
- JSON : JavaScript Object Notation
Fichier CSV
Fichier texte simple pour stocker des données tabulaires.
Les différentes variables sont séparées par ',' ou autre (';', '|', etc)
Première ligne = header (optionnel).
Exemple
"id" "mbikestands" 16104 30.5303867403315 15063 5.19337016574586 17010 16.2651933701657 13045 8.48066298342541 10025 9.31491712707182 ...
Fichier XML ou JSON
Fichier texte structuré pour stocker des données "arborescentes".
Champs séparés par des balises (XML) ou accolades (JSON).
Pas de header : les noms des variables sont répétées dans le fichier.
Exemple
<personnes>
<personne>
<nom>Dupond</nom>
<service>Achats</service>
</personne>
<personne>
<nom>Durand</nom>
<service>Achats</service>
</personne>
<personne>
<nom>Dupuis</nom>
<service>Courrier</service>
</personne>
</personnes>{
"personnes": {
"personne": [
{
"nom": "Dupond",
"service": "Achats"
},
{
"nom": "Durand",
"service": "Achats"
},
{
"nom": "Dupuis",
"service": "Courrier"
}
]
}
}Exemple plus complexe
<CDs>
<CD>
<nom>The Hurting</nom>
<type>Album</type>
<groupe>
<nom>Tears for Fears</nom>
<membres>
<membre>
<nom>Roland Orzabal</nom>
<rôle>Chant</rôle>
</membre>
<membre>
<nom>Curt Smith</nom>
<rôle>Basse</rôle>
</membre>
</membres>
</groupe>
<pistes>
<piste>
<numéro>2</numéro>
<nom>Mad World</nom>
<durée>3:35</durée>
</piste>
...
</pistes>
</CD>
...
</CDs>Encodage
Table ASCII : 128 caractères codés sur 8 bits (1 octet).
| Lettre | Code |
|---|---|
| A | 65 |
| ... | ... |
| Z | 90 |
| a | 97 |
| ... | ... |
| 97 | 122 |
Ok pour un texte en anglais.
...Mais âvèc des åccents ?!
Table ASCII étendue : 256 symboles incluant divers caractères européens - mais pas tous !
$\Rightarrow$ plusieurs codes spécifiques à un groupe de langues.
Unicode
Idée : immense table contenant des codes pour tous les caractères de
toutes les langues connues (parlées ou non !).
$\Rightarrow$ 1,114,112 codes.
Attention : ces codes peuvent être représentés de plusieurs manières.
- Idée simple mais peu économe : toujours utiliser 4 octets (UTF-32).
- Idée opposée : n'utiliser qu'un octet si possible (UTF-8).
- Idée intermédiaire : toujours utiliser au moins deux octets (UTF-16).
...Et il existe plein d'autres encodages.
Jouer avec les encodages en Python
chr(u) : caractère correspondant au point de code Unicode 'u'. Exemple :
>>> chr(70)
'F'
>>> chr(171)
'«'
>>> chr(573)
'Ƚ'
>>> chr(12573)
'ㄝ'encode(e) encode un texte selon 'e'. Exemple :
>>> 'F'.encode("UTF-8")
b'F'
>>> '«'.encode("UTF-8")
b'\xc2\xab'
>>> 'Ƚ'.encode("UTF-8")
b'\xc8\xbd'
>>> 'ㄝ'.encode("UTF-8")
b'\xe3\x84\x9d'Fins de lignes et OS...
$\neq$ operating systems use $\neq$ characters to mark the end of line:
- Unix / Linux / OS X uses LF (line feed, '\n', 0x0A)
- Mac prior to OS X use CR (carriage return, '\r', 0x0D)
- Windows / DOS uses CR+LF (carriage return followed by line feed, '\r\n', 0x0D0A)
Outils pour faciliter la conversion : dos2unix et unix2dos.
...Ou ligne de commande 'tr', 'sed', 'awk' etc.
...Ou ':set ff=unix|dos' avec vim.
Ligne de commande
Obtenir de l'aide sur la commande 'cmd' :
man cmdRedirections en entrée / sortie :
R --no-save --quiet < script.R > sortieEnchaîner des commandes :
wget http://script.R | R --no-saveAfficher un fichier :
head, tail, cat, lessAnalyser un fichier
grep : global regular expression
Filtrer toute les lignes contenant 'tot' :
grep tot fichier.txt
Filtrer toute les lignes contenant un chiffre de 0 à 4 suivi
d'un nombre quelconque de caractères et d'un chiffre de 5 à 9 :
grep '[0-4].*[5-9]' fichier.txtFiltrer toute les lignes commençant par un tiret :
grep '^-' fichier.txtVoir aussi les options -i, -n et -c, ...
Expressions régulières
- . = n'importe quel caractère.
- ^ = début de ligne.
- $ = fin de ligne.
- * = 0...n répétitions.
- + = 1...n répétitions.
- ? = 1 ou 0 répétitions du caratère précédent.
- {m} = exactement $m$ copies du groupe précédent.
- {m,n} = entre $m$ et $n$ copies du groupe précédent.
- [] = classe de caractères. [amP] = a ou m ou P, etc.
Éditer un fichier
Éditeurs classiques : vim, nano.
Outil pratique : sed, "stream editor". Exemples :
# Remplacer toutes les occurrences de "ficheir" par "fichier" :
sed -i 's/ficheir/fichier/g' fichier.txt
# Supprimer toutes les lignes vides :
sed -i '/^[ \t]*$/d' fichier.txt
# Supprimer les lignes 7 à 9 :
sed -i '7,9d' fichier.txtVoir aussi awk (programme similaire), et perl.
Problème d'encodage
file : informations sur un fichier
file -i fichier.pdf
# "application/pdf; charset=binary"
file fichier.pdf
# "PDF document, version 1.5"("mediainfo" est aussi très utile).
iconv : changement d'encodage
# Par exemple iso-8859 → utf8
iconv -f ISO-8859-1 -t UTF-8 fichier.txt -o fichier.utf8Import en R de fichiers type csv
data <- read.table(...)
# ou :
data <- read.csv(...)- ! séparateur de champs
- ! en-tête
- ! conversion de chaîne de caractères en facteur
- ! séparateurs décimaux : , ou .
- ! séparateurs de chaîne de caractères " ou ' ?
Exercice (20 mn)
Importer proprement dans R le fichier exo1.csv qui contient des problèmes d'encodage et de formatage avec read.table.
Vérifier que les variables numériques sont bien numériques, que les chaines de caractères sont bien des chaines de caractères...
Même chose avec read.csv.
Exercice "corrigé" (plusieurs solutions !)
# J'ai commencé par transformer le fichier en dehors de R,
# dans un shell bash. Ce n'est pas nécessaire sous Windows.
vim exo1.csv #visualisation
# Les 5 premières lignes ne sont pas utiles :
sed -i '1,5d' exo1.csv
file exo1.csv
# exo1.csv: ISO-8859 text, with very long lines, with CRLF line terminators
# => on commence par supprimer les \r (Windows --> Linux)
tr -d '\r' < exo1.csv > exo1_.csv
file exo1_.csv
# exo1_.csv: ISO-8859 text, with very long lines
# Reste donc à changer l'encodage (j'utilise toujours UTF-8).
# On peut aussi préciser encoding = "latin1" dans R...
iconv -f ISO-8859-1 -t UTF-8 exo1_.csv -o exo1__.csv
file exo1__.csv
# exo1__.csv: UTF-8 Unicode text, with very long lines : OK !# Séparateur décimal = ','. Conversion des chaînes de caractères en nombres.
data = read.table("exo1__.csv", header=T, sep=",", dec=",", stringsAsFactors=F)
# Vérification
sapply(1:ncol(data), function(i) c(class(data[,i]), data[1,i]))Analyse des fichiers JSON & XML
Package rjson : lecture / écriture de JSON
Lire un fichier JSON :
data <- fromJSON(file="fichier.json")Exporter un objet R en JSON :
json_data <- toJSON(data)Manipuler l'objet JSON :
lapply(data, function(x) { ... }) #ou sapply
unlist(data)
...Exercice (20mn)
Créer la data.frame suivante :
"id" "mbikestands" "1" 16104 30.5303867403315 "2" 15063 5.19337016574586 "3" 17010 16.2651933701657 "4" 13045 8.48066298342541 ...
contenant les id des stations velib et la moyenne du nombre de bornes disponibles sur la période enregistrée
Pour cela vous utiliserez le fichier exo2.json ; structure :
Tableau de stations ayant chacune une id (id) et trois tableaux associés : nombre de vélos (available_bikes), nombre de bornes (available_bike_stands), et date de la mesure (download_date).
Exercice corrigé
library(rjson)
# Attention le premier argument de fromJSON serait une chaîne de caractères.
# Il faut donc bien spécifier file = ...
data = fromJSON(file="exo2.json")
# Méthode 1 : construction des deux vecteurs, puis de la data.frame
means = sapply(data, function(cell) mean(cell$available_bike_stands))
ids = sapply(data, function(cell) cell$id)
df = data.frame(ids=ids, avgbikestand=means)
head(df)
dim(df)
# Méthode 2 : tout en une ligne
df = as.data.frame(t(sapply(data,
function(cell) c(as.integer(cell$id),
mean(cell$available_bike_stands)))))Les fichiers XML
- Fichier texte contenant des balises imbriquées.
- Chaque balise peut être décrite par différents attributs.
- Les balises et attributs devraient être décrits par une dtd et/ou des namespaces.
Parser un fichier
2 méthodes :
- SAX : lecture en ligne + évènements à la lecture d'une balise particulière.
- DOM : construction de l'arbre DOM du document.
Exemple
<?xml version='1.0' encoding='UTF-8'?>
<stations lastUpdate="1409819465886" version="2.0">
<station>
<id>2</id>
<name>Dézery/Ste-Catherine</name>
<terminalName>6002</terminalName>
...
</station>
<station>
<id>3</id>
<name>St-Maurice/St-Henri</name>
<terminalName>6003</terminalName>
...
</station>
</stations>Package XML
- Parse un fichier et construit un arbre DOM.
- Fonctions de parcours et extraction des données de l'arbre créé.
Exemple d'utilisation
data <- xmlTreeParse("exo3.xml") #parse le fichier
xmltop <- xmlRoot(data) #recupère la racine
child <- xmlChildren(xmltop) #les enfants
val <- xmlValue(xmltop) #la valeur
res <- xmlSapply(xmltop, xmlValue) #applique une fonctionExercice (20mn)
Construire à partir du fichier exo3.xml une data.frame contenant les variables suivantes : 'id', 'lat', 'long', 'nbBikes', 'nbEmptyDocks'.
Correction
library(XML)
# Avec xmlTreeParse + fonctions du package XML
data <- xmlTreeParse("exo3.xml") #parse le fichier
stations <- xmlChildren(xmlRoot(data)) #liste des stations
vars <- c('id','lat','long','nbBikes','nbEmptyDocks')
resMatrix <- sapply(stations,function(x) {
# Extraction des variables
clist <- lapply(xmlChildren(x), xmlValue)
# Sélection des variables
sel <- names(clist) %in% vars
# Conversion des variables
as.numeric(unlist(clist[sel]))
})
# Mise sous forme de data.frame
res <- data.frame(t(resMatrix), row.names = NULL)
names(res) <- c('id','lat','long','nbBikes','nbEmptyDocks')
# Avec xmlParse + xmlToList (obtenant une liste R)
exo3 <- XML::xmlParse("exo3.xml")
L = XML::xmlToList(exo3)
length(L) # 452
# L$.attrs == L[[452]] == attributs de la première balise, à ignorer
df = t(sapply(1:451, function(i)
L[[i]][c('id', 'lat', 'long', 'nbBikes', 'nbEmptyDocks')]))
# Ou (équivalent, plus lisible) :
df = t(sapply(L[1:451], function(x)
x[c('id', 'lat', 'long', 'nbBikes', 'nbEmptyDocks')]))R : cheat-sheet.pdf
- Language de haut niveau.
- Support natif des valeur manquantes.
- Programation objet.
- Éco-système vivant : beaucoup de packages.
- ! Plutôt permissif...
- https://cran.r-project.org/doc/contrib/Goulet_introduction_programmation_R.pdf
- ...
Les types de base
Vecteurs :
# vecteur d'entiers (?!)
a = c(1,5,10)
class(a)
# de chaînes de caractères
b = c("a","g","t","t","c","g","g")
class(b)- Permet de stocker des éléments de même type.
- ! Indexation logique
Facteurs :
b = c("a","g","t","t","c","g","g")
c = factor(b,levels=c("a","t","g","c"))
levels(c)
unclass(c)- Type particulier de vecteurs pour coder des catégories : "level(s)"
- ! Interprétation des chaînes de caractères comme des facteurs lors de la création d'une data.frame ; cf option stringAsFactor.
Matrices :
# Matrice d'entiers
a = matrix(c(1,5,10,10),2,2)
# ...de chaînes de caractères
b = rbind(c("a","g"),c("t","t"),c("c","g"),c("t","g"))
c = cbind(c("a","g"),c("t","t"),c("c","g"),c("t","g"))Permet de stocker des éléments de même type dans un tableau.
Arrays :
# Tenseur de dimension 3
a = array(runif(50),dim=c(5,5,2))
a[1,,]
a[,5,]
a[,2,1]
a[2, a[2,,1] < 0.5, ] = 32Permet de stocker des éléments de même type.
Listes :
L = list(a,b,c)
length(L)
L[[2]]
L = list(a=a,b=b,c=c)
L$c ; L[[2]]Permet de stocker des éléments de types différents.
data.frame :
d = data.frame(v1=rep("a",10),v2=1:10,v3=runif(10))
dim(d)
d$v1
d$v4 = factor(rep(c("a","b"),5),levels=c("a","b"))
d[d$v4=="a",]
d[,"v2"]
d[,c(3,1)]
d[,c("v2","v4")]
subset(d, subset=1:3, select=c(v2,v3))
names(d)
summary(d)- Permet de stocker des éléments de types différents.
- = liste de vecteurs nommés indexable et manipulable comme une matrice.
Instruction "with"
with(data, expr, ...): "Evaluate an R expression in an environment constructed from data, possibly modifying (a copy of) the original data."
"This is useful for simplifying calls to modeling functions."
with(mtcars, mpg[cyl == 8 & disp > 350])
# is the same as, but nicer than
mtcars$mpg[mtcars$cyl == 8 & mtcars$disp > 350]
aq <- with(airquality, {
lOzone <- log(Ozone)
Month <- factor(month.abb[Month])
cTemp <- round((Temp - 32) * 5/9, 1) #Fahrenheit to Celsius
data.frame(lOzone, Month, cTemp)
})
head(aq)Fonctions :
f = function(a,b) {
return(a-b)
}
f(5,6)
f(b=5,a=6)
f = function(a=32,b=12){
a-b
}
f()
f(5,6)
f(b=5,a=6)- Une variable comme une autre ?
- Arguments nommés et valeurs par défaut.
- Pas besoin de return explicite.
Vectorisation
! éviter les boucles explicites : préférer les opérations vectorielles.
a=runif(100000)
# Boucle for (bad !)
t=Sys.time()
for (i in 1:length(a)) {a[i]=a[i]+5}
t1=Sys.time()-t
# Vectoriel (good :) )
t=Sys.time()
a=a+5
t2=Sys.time()-t
as.numeric(t1)/as.numeric(t2) #environ 72Beaucoup de fonctions R s'appliquent sur des vecteurs.
Manipulation de données en R
Apply, lapply, sapply
Appliquer une fonction à chaque élément d'un objet.
À préférer aux boucles !
a = data.frame(v1=runif(5000),v2=rnorm(5000),v3=rbinom(5000,5,0.2))
# Appliquer à chaque ligne
r = apply(a,1,sum)
head(r); class(r); dim(r)
# ...ou à chaque colonne
r = apply(a,2,function(col) {c(max(col),which.max(col))})
# Appliquer à tous les éléments d'une liste
b = list(v1=runif(5000),v2=rnorm(5000),v3=rbinom(5000,5,0.2))
r = lapply(b,which.max)
r = sapply(b,which.max)Subset : sample, logical indexing
# Sélectionner une partie des données
a[a$v1>0.98 & a$v3==3,]
# Avec la fonction subset
subset(a,v1>0.98 & v3==3)Binning : cut
# Construire des facteurs par découpage en intervalles
r = cut(a$v2,c(-Inf,-3,-2,2,1,Inf))
class(r); head(r)Jointure : merge, %in%, match
a = data.frame(id=1:500,val1=runif(500))
b = data.frame(id=sample(500,500),val2=runif(500))
# Jointure par colonne de même nom
c = merge(a,b)
# Recherche des indices de correspondances
match(a$id,b$id)[1:10]
# Jointure manuelle
d = cbind(a,b$val2[match(a$id,b$id)])
sum(d!=c)Jointure : suite.
# Matching multiples
b = data.frame(id=sample(500,1000,replace=T),val2=runif(1000))
length(match(a$id,b$id))
sum(!is.na(match(a$id,b$id)))
# match(b$id,a$id) : "full match"
head(a$id %in% b$id) ; length(a$id %in% b$id)
sum(a$id %in% b$id)
c = merge(a,b)
dim(c)
c = merge(a,b,all.x=T)
dim(c)Aggrégation : tapply, by, aggregate
a = data.frame(id=1:500,
val1=runif(500),
val2=factor(rbinom(500,5,0.4)))
aggregate(a$val1,list(a$val2),sum)
tapply(a$val1,list(a$val2),summary)
by(a$val1,list(a$val2),summary)Comptage : table
# Comptage des occurrences de chaque valeur distincte
table(a$val2)
a$val3 = rep(c("a","t","g","c"),500/4)
# Comptage des couples distincts (val2,val3)
table(a[,c('val2','val3')])Exercice
Considérant le "covid dataset", obtenez un jeu de données avec une ligne par pays, contenant les continents, noms de pays, taux de décès ainsi que les taux de vaccination totale et partielle au 31 décembre 2021. Il ne doit plus y avoir de valeurs manquantes.
Affichez ensuite la moyenne du taux de décès et de celui de vaccination partielle par continent (sans utiliser les chiffres par continent déjà existants dans les données). Ces statistiques sont-elles fiables ?
"Corrigé covid"
data <- read.csv("owid-covid-data.csv")
dim(data)
# Une ligne par pays
data <- subset(data, date=="2021-12-31",select=c(
"iso_code","continent","location","total_deaths_per_million",
"people_vaccinated_per_hundred","people_fully_vaccinated_per_hundred"))
# Élimination brutale des lignes avec valeurs manquantes :
data <- na.omit(data)
# Autre stratégie = essayer de compléter (Google...)
# Regroupement par continent + calcul des indicateurs.
# Note : première ligne = statistiques mondiales
aggregate(data[,4:5], list(as.factor(data$continent)), mean)
# A priori peu fiable car continents (très) sous-échantillonnés
# Comparaison avec les chiffres par continent fournis :
data[nchar(data$iso_code) >= 4 & data$continent == "",3:5]Package dplyr
Librarie pour faciliter la manipulation de données.
! Introduction d'un nouvel opérateur
h = function(x) x^3
3 %>% h #= h(3) = 27
f = function(x,y) 2*x + y
g = function(x,y) x * (y+2)
5 %>% f(1) #= f(5, 1) = 11
3 %>% f(2) %>% g(8) #= g(f(3, 2), 8)→ Facilite la lecture du code.
Sélection de lignes
data %>% filter(condition)
data %>% distinct(v1)
data %>% slice_sample(prop=0.3,replace=TRUE)
data %>% slice_max(v1,n=5)
data %>% slice(20:30)
# Possibilité d'enchaîner :
iris %>% filter(Petal.Length < 5)
%>% slice_max(Petal.Width,n=7)Sélection de colonnes
data %>% select(v1,v2)
data %>% select(contains('var'))
data %>% select(-v3)
# ...etc
# Et toujours :
iris %>% filter(Petal.Length < 5)
%>% select(startsWith("Petal"))Transformation de colonnes
data %>% mutate(v3=cumsum(v1/v2))
data %>% rename(v4=v1)
# Voir aussi transmute()
# Composition : transforme toutes les
# colonnes en facteurs sauf le nom
starwars %>% select(name, homeworld, species)
%>% mutate(across(!name, as.factor))Groupement, résumé
data %>% summarise(v1m=mean(v1))
data %>% count(v4)
# ...Peu d'intérêt : simple moyenne.
# Moyenne par groupes :
data %>% group_by(v2)
%>% summarise(v1m=mean(v1))
starwars %>% group_by(hair_color)
%>% summarise(tm = median(mass, na.rm=T))Jointure "X_join"
band_members %>% inner_join(band_instruments)
band_members %>% left_join(band_instruments)
band_members %>% right_join(band_instruments)
band_members %>% full_join(band_instruments)
> band_instruments
name plays
1 John guitar
2 Paul bass
3 Keith guitar
> band_members
name band
1 Mick Stones
2 John Beatles
3 Paul BeatlesVoir aussi...
Le package dplyr fait partie de l'ensemble de librairies tidyverse, écrites dans le même esprit "fonctionnel" :
- ggplot2 : graphes (grammaire graphique).
- tidyr : nettoyage de données.
- readr : lecture de données.
- purrr : map / reduce pour éviter des boucles.
- tibble : variante de la data.frame.
- stringr : opérations sur chaînes de caractères.
- forcats : opérations sur les facteurs.
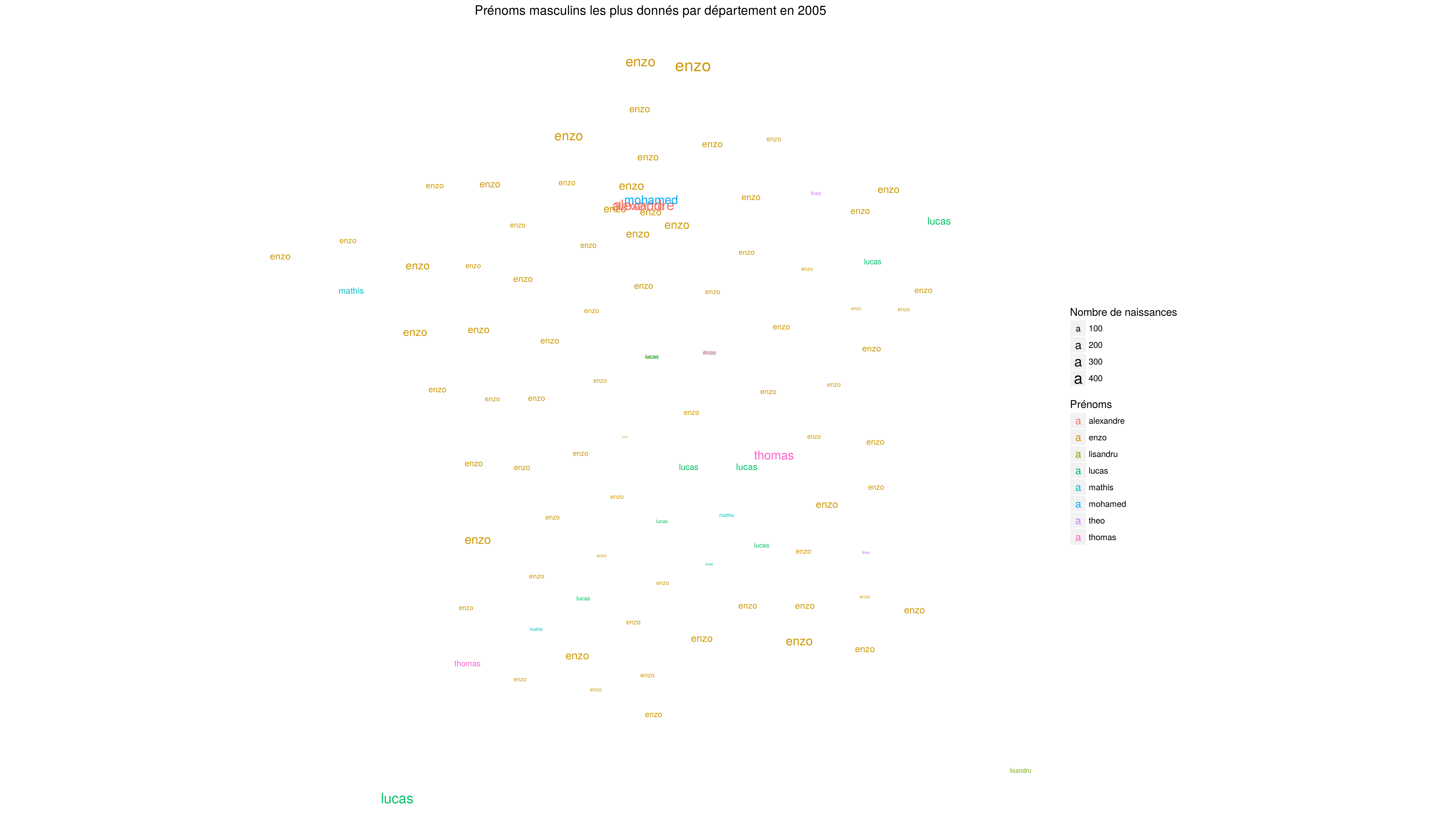
Exercice "prénoms"
Faire une carte représentant les prénoms masculins les plus fréquement donnés aux enfants nés en 2005 pour l'ensemble des départements français. Les données à utiliser sont disponibles dans le répertoire data :
- exo5_dep.csv : centres des départements.
- exo5_prenoms.csv : fichier des prénoms.
Corrigé "prénoms"
departements = read.csv("exo5_dep.csv")
prenoms = read.csv("exo5_prenoms.csv", sep=" ")
plotFreqMap <- function(sexe, annee) { #eg. "M" and "2005"
prenomfreq = prenoms %>% filter(year == annee & sex == sexe)
%>% group_by(dep)
%>% summarise(maxp=name[which.max(n)],
relcount=100*max(n)/sum(n))
geodata = prenomfreq %>% inner_join(departements, by="dep")
plot(geodata$lat, geodata$long, pch=NA)
text(geodata$lat, geodata$long, geodata$maxp, cex=geodata$relcount/4)
}
Avec ggplot2, by Marine D.
# Chargement des data.frame
dep <- read.table("exo5_dep.csv", header = TRUE, sep = ',')
prenom <- read.table("exo5_prenoms.csv", header = TRUE, sep = ' ')
# Jointure pour avoir tout dans un même tableau
join <- prenom %>% left_join(dep)
#View(join)
new_join <- join %>%
filter(year == 2005, sex == 'M') %>%
group_by(dep) %>%
# TODO: on doit pouvoir éviter le quadruple calcul "max(n)"
# en cherchant d'abord ind_max puis en re-summarisant...
summarise(max = max(n), name = name[which.max(n)],
lat = lat[which.max(n)], long = long[which.max(n)])
#View(new_join)
ggplot(data = new_join, aes(lat, long)) +
geom_point(aes(size = max, col = name))