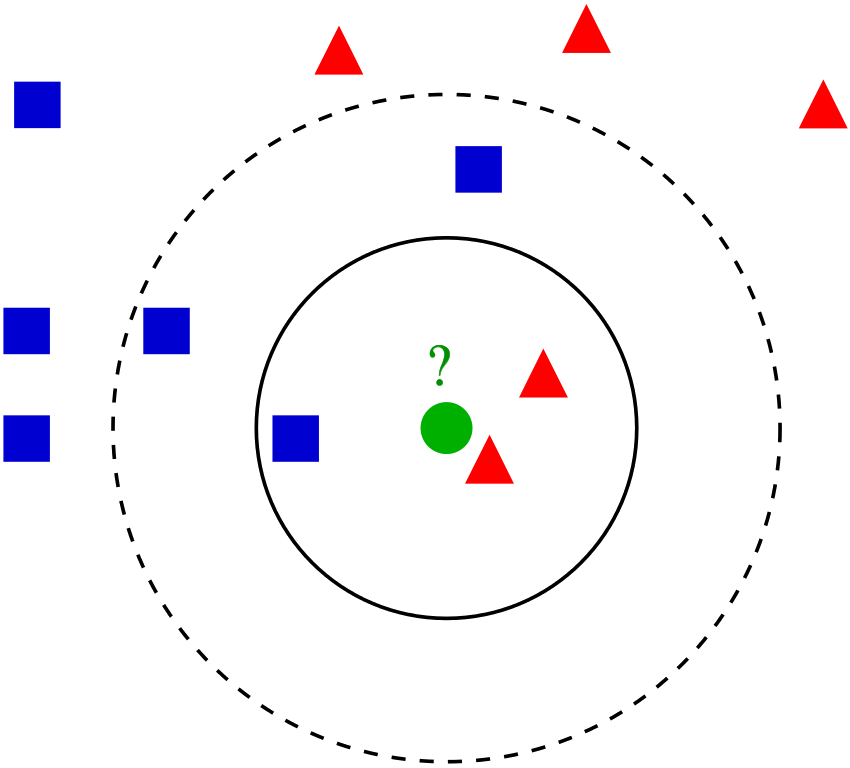
Idée
Un individu devrait ressembler à ses "voisins".
On peut alors prédire...
- sa classe (exemple : pluie) en regardant celles de ses voisins,
- sa valeur (exemple : température) en regardant celles de ses voisins.
Exemple 1
Ciblage publicitaire : montrer au client $X$ des publicités ayant fonctionné sur ses voisins.
Questions :
- quelle distance inter-profils ?
- combien de voisins ?
Exemple 2
Une banque dispose de profils de clients détenteurs d'une carte de crédit :
- âge,
- nombre de personnes à charge,
- ancienneté,
- nombre de produits bancaires, etc
Qui risque de mettre fin à son contrat ?
Méthode (naïve)
Chaque jour,
- comparer les profils clients actifs à ceux ayant clôturé leur compte à $J-x$
- contacter ceux dont les "voisins" sont plutôt des comptes fermés,
en supposant une actualisation journalière de la BD
Avec Python
# Chargement du jeu de données (ou pandas.read_csv(...))
iris = sklearn.datasets.load_iris()
# Définition du modèle : vote majoritaire
# parmi les 15 plus proches voisins.
model = sklearn.neighbors.KNeighborsClassifier(n_neighbors=15)
# "Entraînement" du modèle :
fitted = model.fit(iris["data"], iris["target"])
# Évaluation de l'erreur (sur l'ensemble d'apprentissage)
predictions = fitted.predict(iris["data"])
erreurs = sum(predictions != iris["target"]) #2Problèmes :
- prédiction sur les données d'entraînement ?
- nombre de voisins = 15 ?
Séparation entraînement / test
Problème : plus proche voisin d'un point = lui-même
$\Rightarrow$ aucune erreur !
Solution : évaluer l'erreur sur un autre ensemble.
# Division en ensembles d'apprentissage / test
X_train, X_test, y_train, y_test =
sklearn.model_selection.train_test_split(
iris["data"], iris["target"], test_size=0.3)
# "Entraînement" :
fitted = model.fit(X_train, y_train)
# Évaluation de l'erreur sur l'ensemble de test :
predictions = fitted.predict(X_test)
erreurs = sum(predictions != y_test)Évaluation de l'erreur
L'ensemble de test peut être (par chance)
- très facile à prédire, ou
- très difficile à prédire.
Idée : recommencer $V$ fois, $V \gtrsim 10$
> param_grid = {'n_neighbors':[7, 9, 11, 13, 15, 19, 23]}
> splits = sklearn.model_selection.ShuffleSplit(
n_splits=10, test_size=0.4)
> scv = sklearn.model_selection.GridSearchCV(
sklearn.neighbors.KNeighborsClassifier(),
param_grid, cv=splits, scoring="accuracy")
> c = scv.fit(iris["data"], iris["target"])
> print(c.best_params_)
{'n_neighbors': 11}Alternative
Certains individus peuvent être prédits plusieurs fois, et d'autres jamais. Exemple :
Train Test [1, 2, 3, 4] [5, 6] [1, 3, 4, 6] [2, 5] [2, 4, 5, 6] [1, 3]
L'individu 4 n'est jamais prédit, 5 l'est deux fois.
Solution :
- partition en $k$ parties de taille $n / k$
- pour $i$ allant de 1 à $k$ :
- test sur le bloc $i$,
- entraînement sur les $k-1$ autres
Illustration
> import numpy as np
> X = np.array([[0,0], [0,0], [0,0], [0,0]])
> y = np.array([0, 0, 0, 0])
> kf = sklearn.model_selection.ShuffleSplit(
n_splits=2, test_size=0.5)
> for train_index, test_index in kf.split(X):
> print("TRAIN:", train_index, "TEST:", test_index)
TRAIN: [2 1] TEST: [0 3]
TRAIN: [3 2] TEST: [0 1]0 prédit deux fois, 2 jamais.
> kf = sklearn.model_selection.KFold(n_splits=2)
> for train_index, test_index in kf.split(X):
> print("TRAIN:", train_index, "TEST:", test_index)
TRAIN: [2 3] TEST: [0 1]
TRAIN: [0 1] TEST: [2 3]Chaque individu est prédit exactement une fois.
$k$-fold cross-validation
> splits = sklearn.model_selection.StratifiedKFold(
n_splits=10, shuffle=True)
> scv = sklearn.model_selection.GridSearchCV(
sklearn.neighbors.KNeighborsClassifier(),
param_grid, cv=splits, scoring="accuracy")
> c = scv.fit(iris["data"], iris["target"])
> c.best_params_
{'n_neighbors': 11}Les deux méthodes sont équivalentes ici.
...Ce n'est pas le cas en général !
> c.cv_results_['mean_test_score']
array([0.97, 0.97, 0.98, 0.97, 0.99, 0.98, 0.97])Régression
Idée = remplacer vote majoritaire par moyenne
$\hat y = \frac{1}{k} \sum_{i=1}^{k} y_{n_i}$
avec $n_1, \dots, n_k$ indices des $k$ PPV.
> diab = sklearn.datasets.load_diabetes()
> param_grid = {'n_neighbors':[3, 5, 7, 9, 11]}
> splits = sklearn.model_selection.LeaveOneOut()
> scv = sklearn.model_selection.GridSearchCV(
sklearn.neighbors.KNeighborsRegressor(),
param_grid, cv=splits, scoring="max_error")
> c = scv.fit(diab["data"], diab["target"])
> c.cv_results_['mean_test_score']
array([-49.2, -47.0, -45.9, -45.4, -46.3, -47.0, -47.7])# ...Variabilité de l'erreur :
> 2 * c.cv_results_['std_test_score']
array([80.1, 71.9, 70.3, 68.2, 66.7, 66.1, 64.9])Amélioration, extension
Moyenne pondérée :
$\hat y \propto \sum_{i=1}^{k} \frac{y_{n_i}}{\|x - x_{n_i}\|}$
Généralisation = noyau :
$\hat y \propto \sum_{i=1}^{n} K(\|x - x_i\|) y_i$
Exemple classique :
$K(z) = \exp\left(-\frac{z^2}{\sigma^2}\right)$
(noyau gaussien)
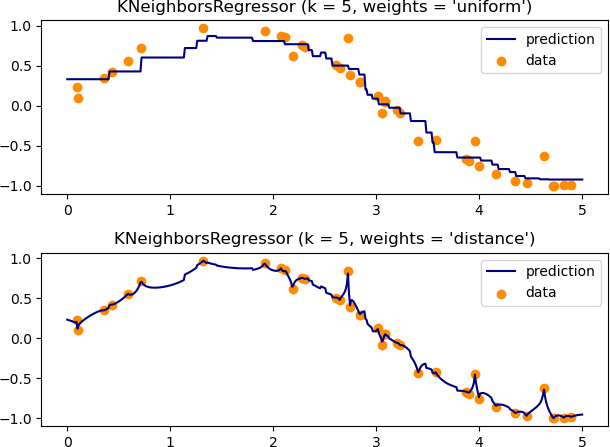
Avec Python
# Pondération par 1 / distance :
scv = sklearn.model_selection.GridSearchCV(
sklearn.neighbors.KNeighborsRegressor(weights='distance'),
param_grid, cv=splits, scoring="max_error")# Noyau gaussien
def kernel(distances):
h = 2 #à ajuster...
weights = np.exp(-distances**2 / h)
return weights
KNeighborsRegressor(n_neighbors=12, weights=kernel)Exercice 0
Reprenez les données iris, et faites varier le nombre de voisins jusqu'à $n-1$. Tracez l'évolution de l'erreur. Expliquez.
Exercice 1
Prenez le temps de comprendre les principaux paramètres de make_regression. Utilisez alors cette fonction pour générer des jeux de données avec 500 lignes et un nombre croissant de colonnes. Comment évoluent les performances du modèle quand n_informative augmente ? Commentez.
Exercice 2
Récupérez les données "Pima Indians Diabetes", disponibles par exemple sur Kaggle.
- Commencez par mettre en forme le jeu de données : valeurs manquantes ? Variables numériques ? Données standardisées ?
- Effectuez alors une validation croisée sur une grille de paramètres.
Exercice 3
Récupérez les données "Iranian Churn Dataset" sur cette page. On cherchera à prédire d'abord "Customer Value" puis "Churn".
- Effectuez une validation croisée sur une grille de paramètres d'abord grossière mais balayant beaucoup de valeurs, puis réduisez la.
- (Régression) Changez les poids pour pondérer avec l'inverse de la distance. Amélioration ?
- (Classification) Évaluez la corrélation entre chaque variable et la sortie.
Ébauche de corrigé
Petit complément
Reprenant ce qui a été dit dans le cours, en expliquant différemment :)
Exercices type examen (TODO: régression ?)
import random, math
import numpy as np
def getData(n, K, rs=None):
x = random.sample(range(0, 10), n)
y = random.sample(range(0, 10), n)
classes = random.choices(range(1, K+1), k=n)
return np.array([x, y]).T, classes
def getTest(n):
return random.sample(range(1, n), math.floor(0.3*n))Appelez "getData(10, 2)" (ou $K=3$), puis calculez le taux de bien classés sur deux ensembles d'entraînement ("getTest(10)").
Recommencez :)